作者👦: 时间👲:2023-12-01 点击数:10
近期,杏盛计算机与信息安全学院人机智能研究室潘细朋博士、刘振丙教授等,联合广东省人民医院(广东省医学科学院)等单位🚵🏿♀️🛄,发表了题为SMILE: Cost-sensitive Multi-task Learning for Nuclear Segmentation and Classification with Imbalanced Annotations的学术论文(https://doi.org/10.1016/j.media.2023.102867),该论文发表在医学图像处理领域TOP期刊《Medical Image Analysis》。潘细朋为论文第一作者,杏盛为论文第一完成单位。《Medical Image Analysis》2022年影响因子10.9👨🏻🔧,JCR分区:Q1😒,中科院分区1区。
该研究针对病理图像分析中细胞核分割与分类标注数据类别分布不均衡、细胞核形态特征多样化等问题🥶,提出了一个类别不均衡标注下的代价敏感多任务细胞核分割与分类模型(SMILE)👲🏿。该研究引入了三个U型网络分支来执行三个任务🦓🫏:细胞核语义分割、实例分割和细胞核分类🛸,为了提高模型的泛化性能👨🏿🎨,提出了一种多任务相关注意力(Multi-task correlation attention🖕🏼,MTCA)机制来促进不同任务之间的特征交互和融合。另外,该研究还提出了一种代价敏感(Cost-sensitive💅,CS)学习策略👩🦳,通过增加对标注类别较少的细胞核被错误分类时的惩罚力度来解决数据不平衡的问题;提出了由粗到精的标记控制分水岭后处理方法🤹🏽🏌🏼♂️,以缓解单个细胞核被过度分割成多个碎片实例的现象🫵🏽。研究结果表明,SMILE在CoNSeP和MoNuSAC 2020数据集上取得了当前最佳性能。病理图像细胞核精准分割与分类有助于病理医生和相关科研人员对肿瘤进行精准诊断🧗🏿♀️、患者疗效评估和预后预测分析🧑🏼🌾。该工作的软件代码和模型已开源,详见链接👳🏻♀️:https://github.com/panxipeng/nuclear_segandcls
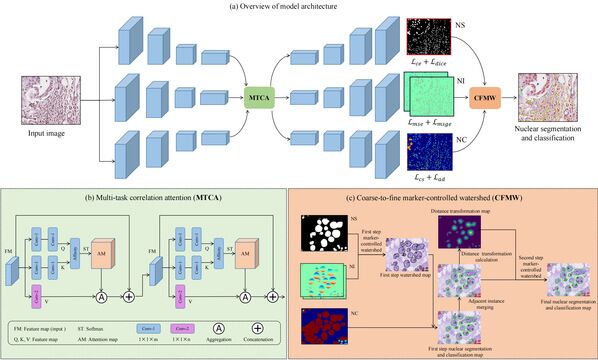
SMILE模型结构示意图
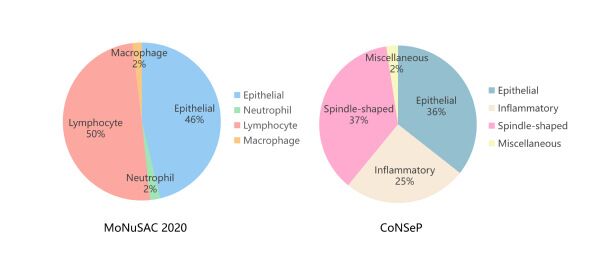
MoNuSAC 2020和CoNSeP不同细胞的比例示意图
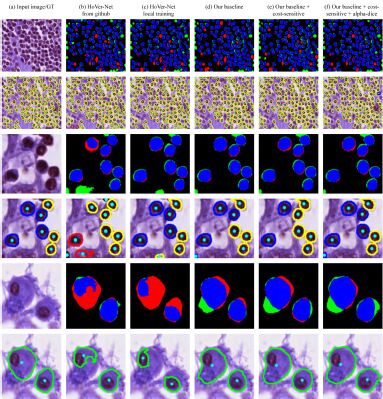
实验的可视化结果
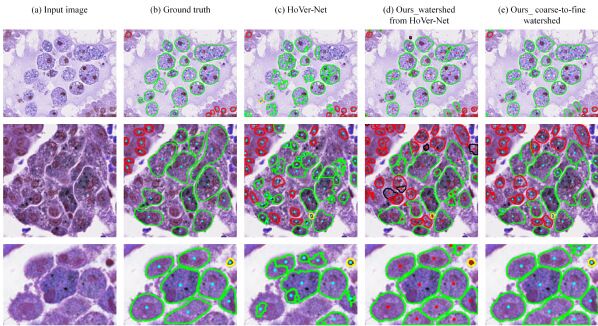
后处理方法的比较分析
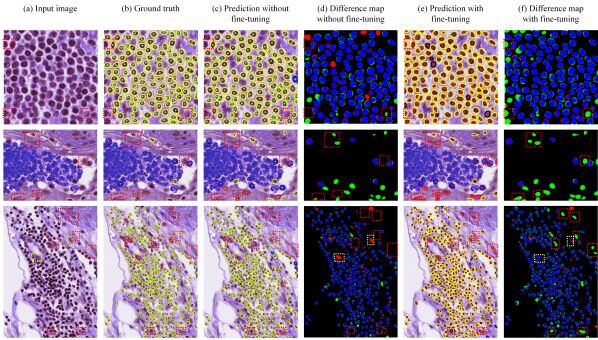
针对不完美注释的fine-tuning可视化
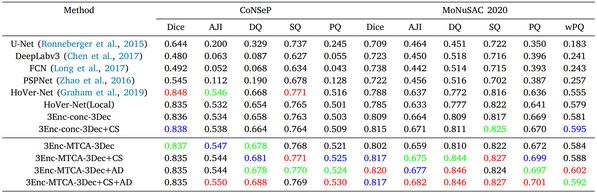
在CoNSeP和MoNuSAC 2020数据集上细胞核分割的实验结果(Top3分别用红色、蓝色和绿色标注)
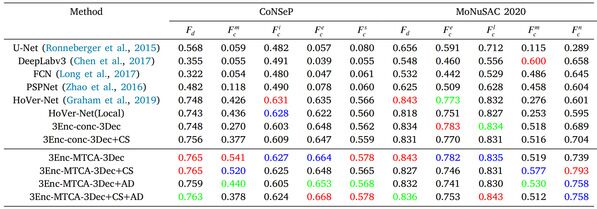
在CoNSeP和MoNuSAC 2020数据集上进行细胞核分类的实验结果